Texto de apropiación científica y tecnológica_____________________________
Módulo 2. Ciclos y genética viral
2.1 ¿Cómo se inicia la infección de una célula
2.2 ¿Cómo entra un virus en una célula
2.3 ¿Cómo se libera el genoma viral en la célula
2.4. Estrategias de replicación de los virus: Adhesión, penetración, desrevestimiento o desnudamiento, replicación, ensamblaje, maduración, liberación
2.5 Virus de ARN de sentido positivo
2.6 Virus de ARN de sentido negativo
2.7 Virus de ARN de doble cadena
2.8 Retrovirus
2.9 Virus de ADN de doble cadena
2.10 Virus de ADN monocatenario
2.11 ¿Qué es la morfogénesis?
2.12 ¿Cómo se liberan los virus de la descendencia?
2.13 Cultivo viral y sistemas de detección directa
________________________________________________________
2.1 ¿Cómo se inicia la infección de una célula?
Como parásitos celulares obligados, los virus no tienen su propio metabolismo; por lo tanto, deben infectar células para reproducirse. Las partículas de virus deben poder reconocer moléculas receptoras específicas en la membrana citoplasmática de la célula huésped y unirse a ellas. Este proceso se conoce como apego. En los virus con envoltura, esta interacción está mediada por proteínas que están incrustadas dentro de la envoltura viral. Este es el caso de los virus de la gripe, así como de los retrovirus y los herpesvirus. La unión de las proteínas de la cubierta viral a las estructuras de la superficie celular es hasta cierto punto muy específica: este es el caso de la interacción entre la proteína de superficie gp120 del virus de la inmunodeficiencia humana (VIH) y el receptor CD4, un polipéptido que se encuentra casi exclusivamente en la membrana citoplasmática de células T auxiliares y macrófagos. En otros casos, las proteínas virales se unen a estructuras celulares que se encuentran en varios tipos de células. Un ejemplo es la unión de la hemaglutinina de los virus de la influenza a residuos de ácido N-acetilneuramínico terminal de oligosacáridos complejos, que se encuentran como modificaciones de proteínas y lípidos en la superficie de la membrana de varias células. En los virus sin envoltura, la superficie de las proteínas de la cápside contiene las estructuras que son responsables de la unión más o menos específica de las partículas a células particulares. Picornavirus, adenovirus y parvovirus son tales ejemplos. Los poliovirus interactúan con un dominio de CD155, una proteína de la superfamilia de las inmunoglobulinas que se localiza en la superficie celular, por el cañón, una estructura similar a una zanja formada por el plegamiento de regiones específicas de aminoácidos de la proteína de la cápside en la superficie de la partícula. La mayoría de los rinovirus, que también pertenecen a la familia de los picornavirus, utilizan la molécula 1 de adhesión intercelular de la proteína de la superficie celular para la unión específica; otros rinovirus se unen a miembros de la familia de receptores de LDL. Los adenovirus se unen al coxsackievirus y al receptor de adenovirus, una proteína de la superficie celular funcionalmente no caracterizada, por el botón al final de las proteínas de fibra, que se encuentran en los vértices de las cápsidas icosaédricas. El receptor se denomina así porque ambos tipos de virus se unen a él. La interacción simultánea de las proteínas adenovirales penton base, también componentes de la superficie de la partícula, con las integrinas avb3 o avb5 también es necesaria para una unión exitosa. Este imperativo de unirse a dos tipos diferentes de receptores para la infección exitosa de las células diana también se encuentra en otros virus, p. en virus de inmunodeficiencia humana y herpesvirus.
2.2 ¿Cómo entra un virus en una célula?
Después de la unión, las partículas de virus que están asociadas con su respectivo receptor en la superficie celular se trasladan al interior de la célula; este proceso se conoce como penetración. En los virus sin envoltura, esto suele realizarse mediante endocitosis mediada por receptores. Las células suelen utilizar este proceso para incorporar moléculas del exterior al citoplasma: los complejos cápside-receptor interactúan con sitios de membrana ricos en clatrina, donde la membrana citoplasmática se invagina alrededor del virus unido, encerrándolo. La vesícula resultante se denomina endosoma. Se invagina hacia adentro, entrando así en el citoplasma de la célula. Las rutas de entrada alternativas son a través de caveolas y caveosomas. Para los próximos pasos del ciclo de infección, las partículas de virus deben liberarse relativamente rápido de los endosomas, porque estos son ricos en proteasas y otras enzimas degradantes, que eventualmente destruirían el virus. Por lo tanto, los virus han desarrollado mecanismos que les permiten salir de la vesícula y evitar el posterior proceso de endocitosis y degradación. Para tales propósitos, los parvovirus poseen actividades enzimáticas similares a la fosfolipasa A2, que como parte de una proteína de la cápside (VP1) son responsables de la liberación del virus del endosoma. Incluso los virus envueltos han desarrollado formas de escapar de la destrucción en los endosomas, ya que también penetran en la célula en parte por endocitosis de vesículas de membrana. Por ejemplo, si los virus de la influenza penetran en una célula, la cápside está rodeada por dos membranas en las vesículas, a saber, la envoltura viral y la membrana vesicular que se deriva de la membrana plasmática. Una secuencia fusionalmente activa de la hemaglutinina viral desencadena la fusión de las dos membranas, lo que conduce a la liberación de la partícula viral de las vesículas. Este proceso se encuentra en variantes similares en muchos otros virus envueltos, p. en flavivirus. La fusión de las dos membranas depende del pH; por lo tanto, la acidificación del interior de la vesícula debe ocurrir previamente. En contraste con este mecanismo de penetración, los paramixovirus poseen en su envoltura una proteína de fusión especial que hace posible, en un proceso independiente del pH, la fusión de la envoltura viral y las membranas celulares durante la unión de la partícula; en este caso, la cápside se libera directamente en el citoplasma después de la fusión de las dos membranas. La fusión de la envoltura viral con la membrana celular también se realiza de manera similar en los herpesvirus y el VIH.
2.3 ¿Cómo se libera el genoma viral en la célula?
La liberación del ácido nucleico viral de la cápside es el resultado de un proceso aún en gran parte sin resolver, que se conoce como decapado. Durante este proceso, el genoma de los virus de ADN (en los herpesvirus también el tegumento) es transportado por diferentes sistemas de transporte intracelular a través de los poros nucleares hacia el núcleo. Una excepción son los poxvirus, que se replican como virus de ADN en el citoplasma de las células infectadas. Después de quitar el revestimiento, el genoma de los virus de ARN permanece como un complejo de proteína y ácido ribonucleico en el citoplasma, donde se producen los siguientes pasos del ciclo de infección. Esta regla solo la rompen los virus de la influenza y el virus de la enfermedad de Borna, que se replican como virus de ARN en el núcleo.
2.4 Estrategias de replicación de los virus
La replicación viral describe los procesos muy complejos de expresión de genes virales y replicación del genoma, que son diferentes en todos los tipos de virus y finalmente dan como resultado la producción de múltiples copias del virus en las células infectadas. Aunque los virus poseen la información genética para la mayoría de los factores necesarios para su propia expresión génica y la replicación del genoma, las proteínas celulares específicas suelen ser esenciales para la expresión génica viral. Estas proteínas suelen actuar como transactivadores de la transcripción de genes virales. Si faltan, el ciclo de infección no puede continuar; el resultado es que no se sintetizan o solo un subconjunto de los productos génicos virales, y la formación de partículas infecciosas permanece incompleta. Esta forma de infección, en la que el virus es capaz de unirse a la superficie de ciertas células y penetrar en ellas, no puede iniciar (o puede iniciar solo parcialmente) el ciclo reproductivo debido a las condiciones intracelulares dadas, se denomina infección abortiva. La dependencia de la replicación del entorno celular es otra razón de la especificidad celular de las infecciones virales. Dependiendo de la naturaleza y estructura del genoma viral, las estrategias de replicación descritas en las siguientes secciones se han encontrado en los diferentes virus.
Un virus debe pasar por el proceso de replicación para crear nuevos viriones infecciosos que puedan infectar otras células del cuerpo o huéspedes posteriores. Después de ingresar al cuerpo, un virus hace contacto físico y cruza la membrana plasmática de una célula objetivo. En el interior, libera y replica su genoma mientras facilita la fabricación de sus proteínas por parte de los ribosomas del huésped. Las partículas de virus se ensamblan a partir de estas moléculas biológicas recién sintetizadas y se convierten en viriones infecciosos. Finalmente, los viriones son liberados de la célula para continuar el proceso de infección.
Las siete etapas de la replicación del virus se clasifican de la siguiente manera:
1. Adhesión
2. Penetración
3. Desrevestimiento o desnudamiento
4. Replicación
5. Ensamblaje
6. Maduración
7. Liberación
Todos los virus deben realizar las siete etapas para crear nuevos viriones. Algunas etapas pueden tener lugar simultáneamente con otras etapas, o algunas etapas pueden tener lugar fuera de orden, según el virus.
1. Adhesión
Una célula interactúa con el mundo extracelular en la membrana plasmática, y es en este lugar donde un virus hace contacto por primera vez con una célula diana, la membrana plasmática de la célula está compuesta por una bicapa de fosfolípidos que tiene numerosas proteínas que sobresalen de la membrana. Estas proteínas de superficie tienen una variedad de funciones que incluyen el transporte de iones y moléculas, facilitando la unión de una célula a otra o actuando como receptores para las proteínas entrantes. La mayoría de las proteínas de la membrana plasmática están glicosiladas, lo que significa que han sido modificadas con azúcares y carbohidratos.
El virus inicia la unión: la unión del virus a la célula huésped. Esta interacción es específica: el virus contiene una proteína de unión al virus que se adsorbe a un receptor de superficie celular en la célula (Cuadro 4.1).
Cuadro 4.1 Receptores de superficie celular para la unión de virus humanos
Virus |
Receptor de superficie celular |
Rhinovirus |
Molécula de adhesión intercelular 1 (ICAM-1) (90 %), receptor de lipoproteínas de baja densidad (10 %) |
Poliovirus |
Receptor poliovirus (PVR) CD155 |
Virus de inmunodeficiencia |
CD4 (receptor); CCR5 or CXCR4 (coreceptores) |
Virus Influenza A |
Ácido siálico |
Virus del sarampión |
CD46, CD150 |
Herpes simplex virus-1 |
Heparán sulfato, HVEM, Nectin-1 |
Virus Dengue |
DC-SIGN |
Las moléculas receptoras diana en la superficie celular son moléculas normales requeridas para las funciones celulares que los virus han evolucionado para explotar, generalmente glicoproteínas o los residuos de azúcar/carbohidrato presentes en las glicoproteínas o la membrana plasmática. Por ejemplo, el rinovirus se une a una proteína conocida como molécula de adhesión intercelular 1 (ICAM-1), involucrada en la unión de una célula a otra. Las cepas del virus de la influenza A se unen a los azúcares de ácido siálico que se encuentran en los extremos de las cadenas de carbohidratos celulares, y los virus del herpes simple (VHS) se unen de manera reversible a los glicosaminoglicanos (GAG), como el heparán sulfato, para unirse a la proteína mediadora de la entrada del herpesvirus o nectinas en la superficie celular (Fig. 4.1).
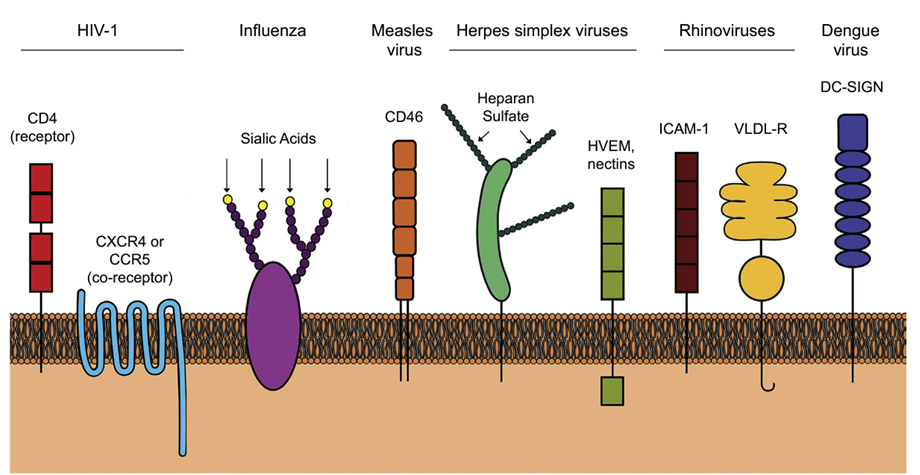
FIGURA 4.1 Receptores de superficie celular. Diferentes virus utilizan receptores de superficie celular específicos para la unión. El VIH-1 requiere CD4 como receptor y receptores de quimiocinas CCR5 o CXCR4 como correceptores. Los virus de la influenza se unen a los residuos terminales de ácido siálico que se encuentran en las glicoproteínas de la superficie celular. Las cepas de laboratorio del virus del sarampión se unen al CD46 (aunque el CD150 también es un receptor del virus). El virus del herpes simple 1 se une inicialmente al sulfato de heparán en los GAG para unirse específicamente a los receptores de entrada, como HVEM o nectinas. El 90% de los rinovirus utilizan ICAM-1 como receptor, mientras que el 10% utiliza el receptor VLDL. El virus del dengue se adhiere mediante DC-SIGN. Tenga en cuenta las diferentes estructuras y tipos de receptores que utilizan los virus para entrar. El tropismo del virus está determinado por qué células del cuerpo expresan el receptor de la superficie celular.
Algunos virus también requieren correceptores para infectar células. El VIH inicialmente se une a una proteína conocida como CD4 en la superficie de los linfocitos T ("células T"), pero requiere una de dos proteínas correceptoras para continuar el proceso de infección., los humanos que tienen una versión modificada de CCR5, uno de estos correceptores, son en gran medida resistente a la infección por el VIH porque el virus no puede utilizar el CCR5 modificado como correceptor y, por lo tanto, se bloquea la infección. La infección de una célula se puede prevenir si se puede inhibir la unión del virus, y las proteínas de unión del virus son el objetivo de muchos fármacos antivirales en uso y desarrollo. La unión implica fuerzas electrostáticas opuestas en la proteína de unión del virus y el receptor de la superficie celular. La proteína de unión del virus se encuentra en la parte más externa del virus, ya que es ahí donde se produce el contacto con la célula. La proteína de unión sobresale de la envoltura de un virus con envoltura, mientras que los virus sin envoltura tienen una o más proteínas de la cápside que interactúan con el receptor de la superficie celular. Las proteínas de unión viral pueden extenderse desde la superficie del virión o pueden estar dentro de “cañones” formados por proteínas de la cápside. Por ejemplo, el 90 % de los serotipos de rinovirus humanos se unen a ICAM-1 en la superficie de las células. En lugar de unirse al exterior de la cápside del rinovirus, la molécula se acopla en un cañón profundo formado por las proteínas VP1, VP2 y VP3 del rinovirus (fig. 4.2A). Por el contrario, el 10% de los rinovirus humanos se adhieren al receptor de lipoproteínas de muy baja densidad (VLDL). Sin embargo, esta interacción no ocurre en los cañones formados por las proteínas virales. En cambio, varias proteínas VP1 en los vértices de la cápside icosaédrica se unen al receptor (fig. 4.2B). Incluso si la afinidad de unión entre la proteína VP1 y el receptor VLDL es baja, las múltiples proteínas VP1 aumentan la fuerza de unión total de la interacción. Este ejemplo también ilustra que diferentes cepas del mismo virus pueden aprovechar diferentes receptores de la superficie celular para unirse.
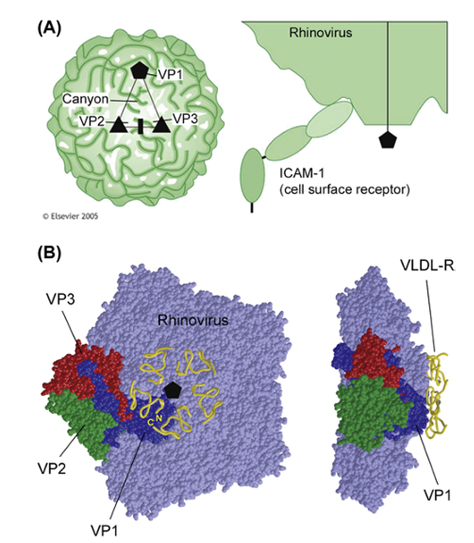
Figura 4.2 Fijación de rinovirus. (A) El 90 % de los serotipos de rinovirus utilizan ICAM-1 como receptor de superficie celular. La proteína celular se une a un cañón formado por las proteínas de la cápside VP1, VP2 y VP3. (B) 10% de los serotipos de rinovirus se unen al receptor de lipoproteínas de muy baja densidad (VLDL-R). A diferencia de la unión de ICAM-1, la unión de estos serotipos de rinovirus se produce en el eje quíntuple en el vértice del icosaedro de la cápside, formado por la repetición de proteínas VP1. Este modelo que llena el espacio muestra la superficie de la cápside del rinovirus (gris) con una unidad estructural resaltada, formada por VP1 (azul), VP2 (verde) y VP3 (rojo). Las moléculas de oro representan los receptores de VLDL y muestran dónde se unen a la proteína VP1 del rinovirus.
2. Penetración
Después de la unión, los virus exitosos ingresan rápidamente a la célula para evitar tensiones extracelulares que podrían eliminar el virión, como el flujo de moco. La penetración se refiere al cruce de la membrana plasmática por el virus. A diferencia de la unión del virus, la penetración requiere energía, aunque la aporta la célula huésped, no el virus. Los virus utilizan varios mecanismos diferentes para entrar en una célula (fig. 4.4, tabla 4.2). Uno de estos aprovecha un proceso normal del huésped: la endocitosis, las células pueden importar moléculas a través del proceso de endocitosis. La endocitosis mediada por receptores se produce cuando los receptores de la superficie celular se unen a sus ligandos y se internalizan en fosas o caveolas recubiertas de clatrina que se convierten en vesículas endocíticas. Con el tiempo, estas vesículas pierden su revestimiento de clatrina o caveolina y se fusionan con "endosomas tempranos", vesículas ligeramente ácidas (pH de 6,0 a 6,5) que se convierten en "endosomas tardíos" a medida que aumenta su acidez (pH de 5,0 a 6,0). Los endosomas tardíos entregan materiales a los lisosomas, vesículas más grandes llenas de enzimas digestivas. Los virus suelen utilizar la endocitosis mediada por receptores para penetrar la membrana plasmática. A medida que desciende el pH del endosoma, las proteínas virales cambian de configuración, lo que les permite escapar del endosoma. Dependiendo del virus, esto puede ocurrir en endosomas tempranos, endosomas tardíos o lisosomas. Tanto los virus envueltos como los no envueltos aprovechan la endocitosis mediada por receptores para entrar en el citoplasma de la célula (fig. 4.4).
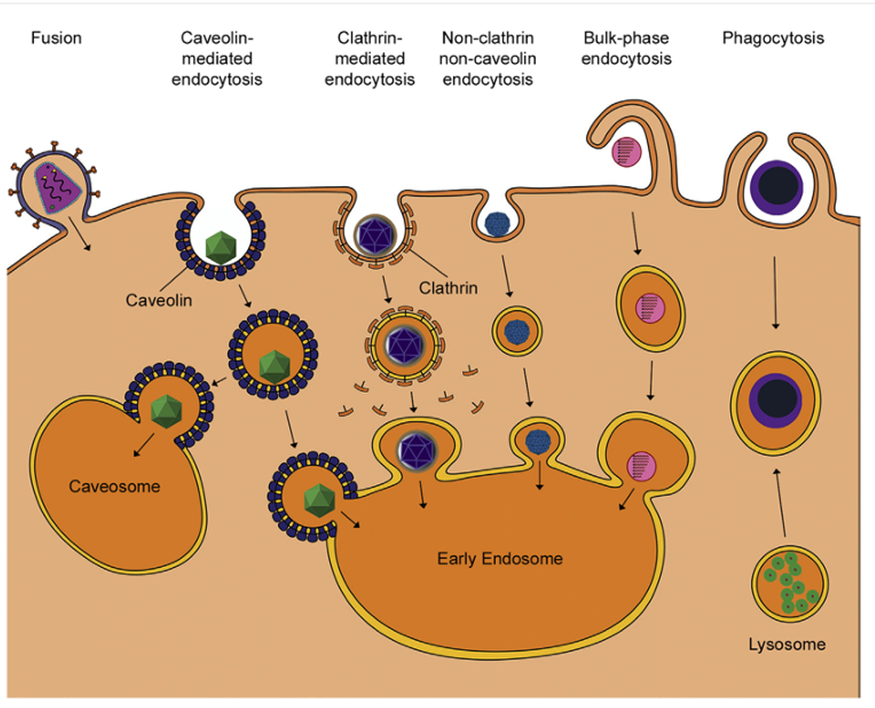
FIGURA 4.4 Penetración viral en la célula. Diferentes virus aprovechan varios mecanismos celulares para ingresar a la célula después de unirse a sus receptores específicos de la superficie celular. Algunos virus envueltos se fusionan, lo que fusiona la envoltura viral con la membrana plasmática. Tanto los virus envueltos como los no envueltos se aprovechan de la endocitosis mediada por receptores en fosas recubiertas de caveolina o clatrina para ingresar al citoplasma de la célula. Otros virus sufren endocitosis mediada por receptor que es independiente tanto de la clatrina como de la caveolina. Los virus también utilizan la endocitosis y la fagocitosis en fase masiva para entrar en la célula.
La mayoría de los tipos de virus usan endocitosis mediada por clatrina para ingresar a la célula, incluidos el virus del dengue, el virus de la hepatitis C y los reovirus. Algunos virus bien conocidos que infectan a los humanos, como el SV40 y los virus del papiloma (que causan verrugas o cáncer de cuello uterino), usan endocitosis mediada por caveolas; esto se descubrió mediante el uso de un fármaco que inhibía la formación de caveolas. El bloqueo de la endocitosis mediada por clatrina no impidió la entrada de estos virus en las células. Otros virus sufren endocitosis mediada por receptor que es independiente tanto de la clatrina como de la caveolina. Los virus también aprovechan otras formas de endocitosis, como la endocitosis en fase masiva y la fagocitosis, para entrar en la célula. En la endocitosis de fase masiva, la célula forma una vesícula que engulle cualquier molécula presente en el líquido extracelular, incluidos los virus. La fagocitosis es una forma de endocitosis mediada por receptores que utilizan células especializadas para engullir células enteras. Recientemente, se demostró que dos grandes virus de ADN, HSV-1 y mimivirus, ingresan a las células a través de vías similares a la fagocitosis.
Un método de penetración que utilizan exclusivamente los virus envueltos es la fusión. La fusión de la envoltura viral puede ocurrir en la membrana celular o dentro de vesículas endocitadas, como el endosoma, y está mediada por la misma proteína viral que usa el virus para unirse o por una proteína viral diferente, según el virus. Por ejemplo, el VIH tiene una proteína conocida como gp120 que se une a CD4 y uno de los dos correceptores de entrada, CCR5 o CXCR4. Una vez que esto ocurre, una proteína viral diferente, gp41, fusiona la envoltura del virus con la membrana celular, liberando la nucleocápside en el citoplasma. Descanso para el estudio Describa las diferentes formas en que los virus pueden ingresar al citosol.
3. Desrevestimiento o desnudamiento
El desrevestimiento se refiere a la ruptura o eliminación de la cápside, lo que provoca la liberación del genoma del virus en la célula, donde se llevará a cabo la replicación y la transcripción del genoma. La eliminación de la cubierta puede estar separada o estrechamente vinculada con la penetración, y los virus logran la eliminación de la cubierta de varias maneras diferentes (Fig. 4.5).
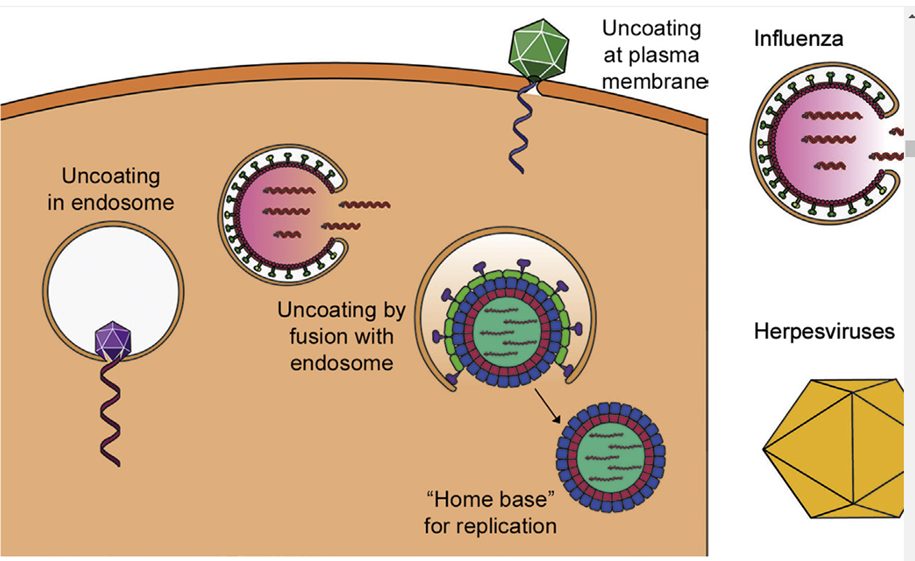
FIGURA 4.5 Eliminación de la cubierta de las cápsides de los viriones. Ciertos virus, incluidos los rinovirus, se expanden para formar poros en el endosoma a través de los cuales puede escapar el genoma viral. Al igual que el virus de la influenza, otros virus inducen la fusión de la envoltura del virión con la membrana endosomal, liberando el genoma viral. Históricamente, se ha pensado que las cápsidas del poliovirus no ingresan a la célula en absoluto: la unión de la cápside al receptor de la superficie celular induce un cambio de conformación que crea un poro en la membrana a través del cual se transporta el genoma. Muchos virus mantienen una cápside parcialmente intacta en el citosol que actúa como una "base de operaciones" para la replicación, como lo hacen los reovirus.
Por ejemplo, los rinovirus ingresan a la célula mediante endocitosis mediada por receptor en vesículas recubiertas de clatrina. Dentro del endosoma ácido, el virus se expande en tamaño alrededor del 4%, y una de las proteínas de la cápside, VP1 (viral proteína 1), forma poros en el endosoma que permiten la liberación del genoma de ARN del rinovirus. Por otro lado, el virus de la influenza tiene una proteína viral conocida como hemaglutinina (HA) incrustada en la envoltura del virus. HA se une a los residuos de ácido siálico que se encuentran en la superficie de las células epiteliales respiratorias y la penetración se produce a través de endocitosis mediada por receptores. El bajo pH del endosoma provoca un cambio conformacional en la proteína viral HA, revelando un péptido de fusión que acerca las dos membranas y fusiona la envoltura viral con la membrana endosomal. En este caso, la proteína HA facilita tanto la unión como el desprendimiento del virus. Los segmentos del genoma de ARN viral liberados se transportan al núcleo y entran a través de los poros nucleares. Se ha pensado que otras cápsides virales, como las del poliovirus, no ingresan a la célula en absoluto: la unión de la cápside del poliovirus al receptor de la superficie celular provoca un cambio conformacional en el virión que crea un poro en la membrana celular a través del cual el El ARN viral se libera en el citoplasma. Por el contrario, muchos virus permanecen prácticamente intactos después de la penetración. Los reovirus no se eliminan por completo dentro del citoplasma, lo que proporciona una "base de operaciones" para la replicación del genoma. Muchos herpesvirus infectan las neuronas, pero deben replicarse en el núcleo, que puede estar bastante lejos de su sitio de entrada en la membrana plasmática. Después de la fusión de la envoltura viral con la membrana plasmática, las nucleocápsides intactas del HSV-1 se transportan a lo largo de los microtúbulos hasta el núcleo. Las proteínas del HSV se unen a la dineína, una proteína de la célula huésped que "camina" vesículas de carga a lo largo de los microtúbulos. En el núcleo, la cápside del VHS se acopla en un poro nuclear y su ADN viral se transporta al núcleo (fig. 4.6).
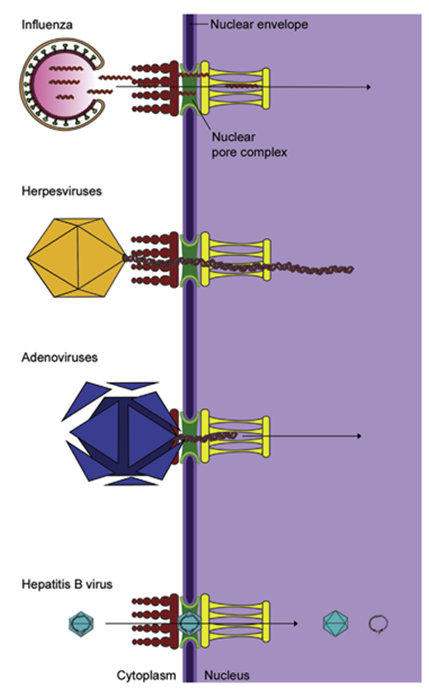
FIGURA 4.6 Transporte de genomas virales al núcleo. Varios virus deben transportar sus genomas al núcleo para que se produzca la transcripción y/o replicación viral. Los segmentos del genoma de la influenza se transportan a través del poro nuclear hacia el núcleo. Las cápsides del herpesvirus se transportan a lo largo de los microtúbulos hasta el poro nuclear, donde se produce el desprendimiento. Las cápsides de los adenovirus se desensamblan en el poro nuclear y el ADN viral se transporta al núcleo. Otros virus, incluido el virus de la hepatitis B, son lo suficientemente pequeños como para que toda la cápside pueda pasar a través del poro nuclear.
Aún otras cápsides son lo suficientemente pequeñas como para pasar a través de los poros nucleares: la cápside de la hepatitis B, con un diámetro de alrededor de 30 nm, puede importarse intacta a través de un poro nuclear para descubrir el interior del núcleo.
4. Replicación
De la misma manera que nuestro ADN codifica la información para fabricar nuestras proteínas, el genoma de un virus actúa como instrucciones para la síntesis de proteínas virales. Para crear nuevos viriones, las proteínas que se incorporarán al virión se fabrican mediante la expresión de genes virales y el genoma del virus se copia mediante el proceso de replicación. La estrategia de replicación de un virus generalmente depende del tipo de genoma de ácido nucleico que contiene (Fig. 4.7). Muchas clases de virus aprovechan las proteínas celulares para replicar sus genomas. Por lo tanto, la ubicación de estas proteínas generalmente dictará dónde tendrá lugar la replicación del ácido nucleico viral. Todos los organismos vivos tienen genomas compuestos de ADN de doble cadena (ds), pero los virus son únicos en el sentido de que sus genomas pueden estar hechos de ADN de una sola cadena (ss), así como dsRNA o ssRNA, que pueden ser de sentido positivo o negativo. El sistema de clasificación de Baltimore, "Estructura y clasificación de virus", es útil cuando se analizan las estrategias generales de replicación de los virus porque clasifica los virus en siete clases según su tipo de genoma:
1. Virus de ADN de doble cadena
2. Virus de ADN monocatenario
3. Virus de ARN de doble cadena
4. Virus de ARN de sentido positivo
5. Virus de ARN de sentido negativo
6. Virus de ARN de transcripción inversa
7. Virus de ADN de transcripción inversa
Los ácidos nucleicos virales se encuentran en una variedad de configuraciones. Pueden ser lineales o circulares, y pueden estar segmentados en varias piezas más pequeñas dentro del virión, como ocurre con los virus de la influenza, o no segmentados como el virus de la rabia, que contienen una molécula de ácido nucleico que codifica todos los genes necesarios. Las moléculas más largas están más sujetas a roturas, pero los virus segmentados deben empaquetar todos los segmentos del genoma en un virión para que sea infeccioso. Independientemente de la estructura de su ácido nucleico, todos los virus necesitan expresar sus proteínas virales y replicar su genoma dentro de la célula para crear nuevos viriones.
2.5 Virus de ARN de sentido positivo
Los genomas de ARN de los virus de ARN de sentido positivo tienen la polaridad de un ARN mensajero (ARNm) y pueden traducirse directamente en proteínas mediante el uso de la maquinaria de traducción celular. Esto da como resultado la síntesis de una gran poliproteína precursora en picornavirus y flavivirus, mientras que en calicivirus, togavirus, arterivirus y coronavirus se encuentran dos o más formas diferentes de las proteínas precursoras. Se escinden proteolíticamente en las proteínas y enzimas estructurales virales. Una de las enzimas es muy importante para la replicación del genoma, a saber, la ARN polimerasa dependiente de ARN. Dado que esta enzima no existe en las células eucariotas, el virus debe codificar él mismo la información genética correspondiente. Usando el genoma de ARN de sentido positivo como molde, esta polimerasa cataliza la síntesis de un ARN de sentido negativo complementario, que a su vez sirve como molde para la producción de un gran número de nuevos genomas de ARN con polaridad de sentido positivo. Prototipos importantes de virus de ARN de sentido positivo son picornavirus, flavivirus, togavirus y coronavirus.
2.6. Virus de ARN de sentido negativo
A diferencia de los virus de ARN de sentido positivo, el genoma de los virus de ARN de sentido negativo no muestra la polaridad del ARNm; por lo tanto, no puede traducirse directamente en proteínas. Esto requiere la transcripción del genoma en una molécula de ARN complementaria, un proceso que se basa en la presencia de una ARN polimerasa dependiente de ARN. Dado que las proteínas virales no pueden sintetizarse directamente a partir del genoma debido a su polaridad de sentido negativo, los virus de ARN de sentido negativo deben llevar esta polimerasa al interior de la célula como parte de la partícula viral. La enzima sintetiza moléculas de ARNm complementarias, que el uso eficiente de la capacidad de codificación se denomina orientación ambisense. También se encuentra en el circovirus porcino, que tiene un genoma de ADN circular monocatenario.
2.7 Virus de ARN de doble cadena
Los reovirus y birnavirus tienen un genoma de ARN segmentado de doble cadena. En este caso, una polimerasa de ARN dependiente de ARN también se encuentra como parte de las partículas del virus y se transporta a la célula durante la infección. Transcribe los fragmentos genómicos de sentido negativo en moléculas de ARNm traducibles cubiertas. Estos también sirven como moldes para la síntesis de nuevas cadenas dobles. Solo los reovirus y los birnavirus siguen este principio de replicación conservadora, en el que ninguna de las cadenas originales está presente en las moléculas de ARN de doble cadena recién sintetizadas.
2.8 Retrovirus
Estos virus de ARN monocatenario tienen un genoma de sentido positivo, pero su ciclo de replicación difiere completamente del de las familias de virus mencionadas anteriormente. Los retrovirus encierran la enzima transcriptasa inversa en sus viriones, y esta se introduce en la célula durante la infección. La actividad de la polimerasa de ADN dependiente de ARN de la transcriptasa inversa cataliza la transcripción de la plantilla del genoma de ARN en ADN de doble cadena, que posteriormente se integra en el genoma celular. Este llamado provirus se comporta como una región cromosómica celular normal; se replica junto con el genoma celular durante la división celular y se transmite a las células hijas. La transcripción y la traducción ocurren solo a partir del ADN viral integrado. Esto produce moléculas de ARNm empalmadas y no empalmadas que se traducen en enzimas y proteínas estructurales virales. La secuencia no empalmada del provirus que abarca todo el ARNm sirve como un genoma viral, que se empaqueta en partículas de virus.
2.10 Virus de ADN de doble cadena
El genoma de estos virus es transcrito por enzimas celulares después del transporte al núcleo. Las moléculas de ARN resultantes se traducen luego en las proteínas estructurales y no estructurales virales. Los hepadnavirus tienen un genoma de ADN parcialmente bicatenario, que se completa en las células infectadas y está presente en el núcleo como una molécula circular. Los hepadnavirus tienen una transcriptasa inversa, lo que enfatiza, junto con algunos otros atributos, su relación con los retrovirus. Durante el ciclo de replicación, esta enzima viral transcribe en ADN el ARNm que abarca todo el genoma. Los virus de ADN más pequeños, como los poliomavirus, no codifican su propia ADN polimerasa, sino que codifican polipéptidos que interactúan con las ADN polimerasas celulares y alteran la función de estas de tal manera que las secuencias de ADN viral se replican preferentemente. Este proceso comienza en el origen de la replicación y procede de forma bidireccional y semiconservadora; es muy similar a la replicación delta o plásmido, que se encuentra durante la replicación de cromosomas bacterianos circulares o moléculas de ADN episomal. Los virus de ADN más complejos, como los adenovirus y los herpesvirus, tienen un patrón de expresión génica estrechamente regulado que se divide en fases temprana y tardía; estos virus también utilizan la maquinaria de transcripción y traducción celular. Varios polipéptidos reguladores y enzimáticamente activos se sintetizan tempranamente, incluidas las ADN polimerasas virales y algunas enzimas que están involucradas en el metabolismo de los ácidos nucleicos, que hacen posible la replicación de los genomas de ADN de doble cadena. Los genomas lineales de los adenovirus se replican en un modo semiconservador; esto significa que cada hebra principal se usa como molde y permanece como parte de las moléculas de ADN de doble hebra recién sintetizadas. Los orígenes de replicación se encuentran en los extremos del ADN de doble cadena. Los genomas de ADN lineal de los herpesvirus están circularizados en la célula. Estos virus pueden tener dos ciclos de replicación diferentes: durante la latencia, el ADN viral está presente como un episoma y es replicado por ADN polimerasas celulares. En el ciclo de infección lítica, que conduce a la producción de descendientes de virus, la replicación ocurre de acuerdo con el principio de replicación sigma, que también ocurre en algunos bacteriófagos, y se denomina replicación en círculo rodante. En este modo de replicación, una cadena de la molécula de ADN circular se escinde en el origen de la replicación, generando un extremo 30-OH libre, que se extiende continuamente mediante la polimerización de más nucleótidos por la ADN polimerasa viral, por lo que la cadena de ADN intacta sirve como una plantilla. El extremo 50 se desplaza continuamente de la hebra plantilla, como si estuviera enrollado. De esta forma, se genera una cadena única de ADN que engloba múltiples copias del genoma del herpesvirus en forma concatemérica, es decir, repetidas en serie. Se convierte en una cadena doble de ADN mediante la síntesis discontinua de fragmentos de Okazaki y se escinde mediante endonucleasas que resuelven los concatémeros en genomas virales individuales. En ambos adenovirus y herpesvirus, la síntesis de proteínas estructurales virales se induce solo en la fase tardía de la expresión génica después de la replicación del ADN. Los poxvirus también son virus de ADN de doble cadena, pero siguen un modo de replicación completamente diferente. Realizan todas las reacciones de síntesis en el citoplasma de las células infectadas. Por lo tanto, todas las enzimas que normalmente se localizan en el núcleo no pueden ser utilizadas por los poxvirus. Estos incluyen polimerasas de ARN, enzimas de protección y enzimas modificadoras de ARN. Por lo tanto, los poxvirus poseen, además de su propia ADN polimerasa, también la información genética para estas funciones. La expresión génica y la replicación del genoma también están estrictamente reguladas en los poxvirus. La familia de los asfarvirus incluye solo un agente patógeno animal: el virus de la peste porcina africana. Los asfarvirus tienen un genoma de ADN de doble cadena y son similares a los iridovirus fitopatógenos en muchos aspectos. La replicación ocurre en el núcleo de las células infectadas.
2.10 Virus de ADN monocatenario
Las familias de parvovirus, anellovirus y circovirus abarcan virus con un genoma de ADN monocatenario lineal o circular. Las tres familias no codifican una ADN polimerasa viral; al igual que los poliomavirus, también utilizan enzimas celulares para la replicación del genoma, y estas se modifican funcionalmente. De esta forma, se generan intermedios de ADN bicatenario complementarios que posteriormente se transcriben en genomas monocatenarios.
5. Maduración
Después de que el genoma del ácido nucleico y otras proteínas esenciales se empaquetan dentro de la cápside, que se ensambló a partir de una o varias proteínas virales traducidas, ocurren los pasos finales de la replicación del virus: maduración y liberación. Hasta este punto, el virión había estado en proceso de formación, y si la célula se abriera en este punto, los viriones no podrían iniciar la infección de nuevas células. La maduración se refiere a los cambios finales dentro de un virión inmaduro que dan como resultado una partícula viral infecciosa. Los cambios estructurales de la cápside a menudo están involucrados, y estos pueden estar mediados por enzimas del huésped o enzimas codificadas por virus. Un buen ejemplo involucra la proteína influenza HA. Participa en la unión al ácido siálico de la célula, como se describió anteriormente, y la proteína HA puede unirse al ácido siálico después de ser glicosilada (mediante modificación postraduccional). Sin embargo, la proteína HA debe escindirse en dos porciones, HA1 y HA2, para volverse infeccioso, porque aunque la porción HA1 se une al receptor de la superficie celular, la porción HA2 es la que fusiona la envoltura viral con la membrana endosómica para liberar el virus en el citoplasma. Esta escisión de HA en HA1 y HA2 se lleva a cabo mediante proteasas celulares (enzimas que escinden proteínas). Por el contrario, la partícula central del VIH está compuesta por proteínas codificadas por el gen gag. El gen se traduce en una poliproteína que es escindida por la proteasa viral para formar las proteínas de la cápside, la matriz y la nucleocápside del virión. En este caso, la maduración se produce después de que el virión se haya liberado de la superficie celular (fig. 4.17) y se requiere para formar un virión infeccioso.
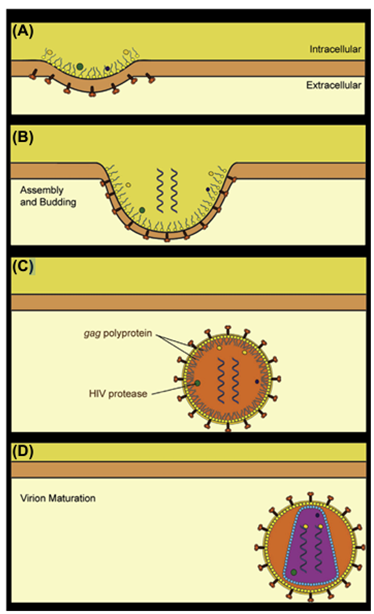
FIGURA 4.17 Montaje, liberación y maduración de viriones de VIH. (A) Las proteínas del VIH se congregan en la membrana plasmática de la célula, lo que hace que se forme un brote en la membrana. El genoma de ARN diploide se empaqueta en la cápside de ensamblaje (B). El virus se libera de la membrana celular, pero la poliproteína Gag aún no se ha escindido para separar las proteínas de la cápside y la matriz del virión (C). La proteasa del VIH escinde la poliproteína, lo que permite que las proteínas completen la arquitectura del virión infeccioso (D).
6. Ensamblaje
Los virus se crean a partir de componentes recién sintetizados y, para ser liberados de la célula, esos componentes deben recolectarse en un sitio particular de la célula y someterse a ensamblaje para formar una partícula de virus inmadura. De la misma manera que la penetración y el desprendimiento son difíciles de separar en el ciclo de algunos virus, el ensamblaje a menudo puede ocurrir junto con la maduración y la liberación. La ubicación del ensamblaje del virión dependerá del virus en particular. Puede tener lugar dentro del núcleo de la célula, en la membrana plasmática o en una variedad de membranas intracelulares, como el complejo de Golgi. La mayoría de los virus de ADN sin envoltura ensamblan su nucleocápside en el núcleo, ya que ese es el sitio de replicación del genoma. Las proteínas virales se importan a través de los poros nucleares para llegar al sitio de ensamblaje. Sin embargo, cuando se ensamblan, la mayoría de los virus de ADN son demasiado grandes para pasar a través de los poros nucleares. En este punto, algunos virus pueden atravesar la envoltura nuclear de doble membrana, mientras que otros inducen la lisis celular o la apoptosis para escapar del núcleo. Por otro lado, los virus con envolturas derivadas de la membrana plasmática suelen ensamblarse allí. El genoma de ácido nucleico de un virus helicoidal está protegido por la repetición de proteínas de la cápside. Debido a esto, las proteínas de la cápside pueden comenzar a envolver el genoma tan pronto como se copia (o viceversa, según el virus: el genoma se puede envolver alrededor de las proteínas de la cápside; Fig. 4.16).
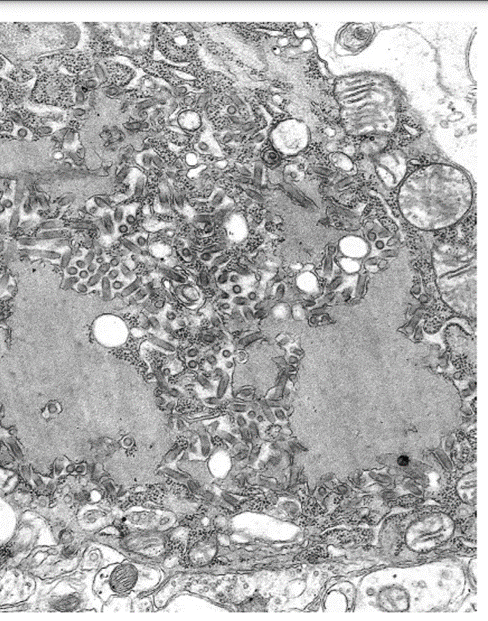
FIGURA 4.16 Asamblea de viriones de rabia. Esta micrografía electrónica de una célula infectada con el virus de la rabia muestra las cápsides en forma de bala ensambladas dentro del citoplasma de la célula. Posteriormente, el virus de la rabia brota de la membrana plasmática.
El ensamblaje de virus con una arquitectura más compleja está orquestado por una variedad de proteínas chaperonas virales llamadas proteínas de andamiaje. Los herpesvirus y los adenovirus son ejemplos de grandes virus icosaédricos que se ensamblan con la ayuda de proteínas de andamiaje.
7. Liberación
El último paso en el ciclo de replicación del virus es la liberación del virión al entorno extracelular, donde puede continuar el ciclo de infección con nuevas células. La liberación puede ocurrir de varias maneras diferentes, dependiendo del virus. Los virus que obtienen su envoltura de la membrana plasmática generalmente se ensamblan en la capa interior de la membrana plasmática, incrustando las proteínas de su envoltura en la membrana plasmática. A medida que las proteínas de la cápside viral interactúan, las proteínas virales asociadas a la membrana hacen que la membrana plasmática comience a curvarse alrededor de la cápside. Esto continúa hasta que la membrana plasmática envuelve completamente al virus, que sale de la célula. Este proceso se conoce como gemación (Fig. 4.17A y B; Fig. 4.18). Los virus pueden brotar de cualquiera de los sistemas de membrana dentro de la célula, incluido el rER, el complejo de Golgi o incluso la envoltura nuclear. En este caso, el virión ya envuelto no necesita brotar a través de la membrana plasmática. Generalmente sufre exocitosis para salir de la célula. Los virus sin envoltura también pueden salir de la célula por exocitosis. Los virus líticos, sin embargo, alteran la membrana plasmática y provocan la lisis o ruptura de la célula. Esto libera los viriones nacientes para infectar nuevas células. Muchos virus humanos sin envoltura se liberan a través de la lisis celular. Los procesos de ensamblaje, maduración y liberación están estrechamente relacionados, pero todos son necesarios para crear una progenie de viriones infecciosos capaces de continuar el ciclo de infección.
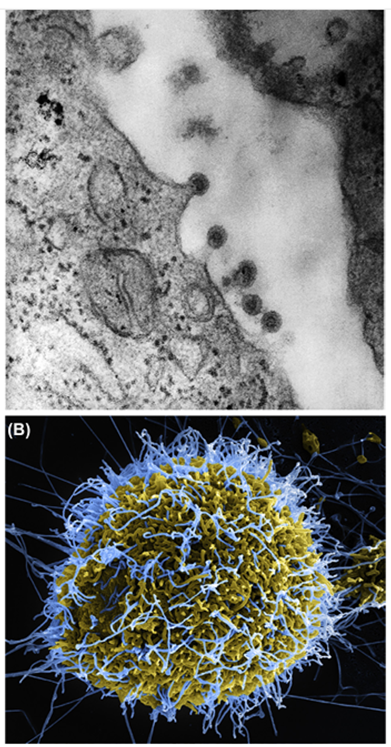
FIGURA 4.18 Germinación de virión. (A) Los viriones del virus de la rubéola se observan brotando de la membrana plasmática del huésped en esta micrografía electrónica de transmisión. (Imagen cortesía de CDC/Dr. Fred Murphy y Sylvia Whitfield.) (B) En esta micrografía electrónica de barrido pseudocoloreada mejorada digitalmente, los viriones helicoidales del Ebolavirus (azul) brotan de una célula infectada (amarillo).
2.13 Cultivo viral y sistemas de detección directa
Para el cultivo y la propagación de la mayoría de los virus, hoy en día se dispone de líneas celulares en continuo crecimiento. El material del paciente preferentemente estéril a investigar, como sangre, suero, lavado faríngeo u orina, se limpia de impurezas crudas y se incuba con las células en un volumen pequeño. Después de la unión de las partículas de virus a las células, se agrega un medio que contiene antibiótico a los cultivos para evitar el crecimiento de bacterias que pueden estar presentes como contaminación en el material de prueba. En los días siguientes se revisan microscópicamente las células en busca de cambios morfológicos, como la aparición de efectos citopáticos, placas en la capa celular por muerte celular, cuerpos de inclusión y células gigantes, que permiten una primera inferencia sobre el virus replicante, y también sirven como evidencia de que los virus infecciosos estaban presentes en el material de partida. La forma clásica de cultivo viral generalmente implica un período de incubación relativamente largo de 1 a 4 semanas. Una versión abreviada es el ensayo de vial de concha. En este método, las células susceptibles se incuban con el material que contiene el virus sospechoso en los pocillos de una microtitulación, una placa de 24 pocillos o sobre portaobjetos de vidrio en tubos de centrífuga y se centrifugan a baja velocidad. Esto probablemente altera la fluidez de la membrana citoplasmática de las células, lo que conduce a una penetración más rápida de los patógenos. Después de la incubación durante 1 o 2 días, las proteínas virales pueden demostrarse en las células mediante inmunofluorescencia o procedimientos similares. Actualmente, las líneas celulares primarias apenas se utilizan para el cultivo de virus patógenos humanos. Solo tienen una capacidad de división limitada; por lo tanto, deben establecerse repetidamente. Una excepción son los fibroblastos del prepucio, que se utilizan ocasionalmente para el cultivo del virus del herpes simple. En particular, el uso rutinario de células madre embrionarias, que poseen una mayor capacidad de división, está muy regulado. Sin embargo, para otras células humanas primarias también debe demostrarse que están libres de virus después del cultivo, en particular, no deben contener virus de hepatitis crónica (B, C, D) o virus de inmunodeficiencia humana (VIH). En medicina veterinaria, las líneas celulares primarias se utilizan comúnmente en casos excepcionales para el cultivo de ciertos virus patógenos en virología avícola. En ciertos casos, incluso se utilizan para la producción de vacunas. Este es el caso, por ejemplo, del parvovirus porcino. El cultivo viral anteriormente generalizado en huevos de pollo embrionados ya no se usa de forma rutinaria. Solo se usa en ciertos casos, como el cultivo de nuevos aislados del virus de la influenza o para la producción de vacunas. Mediante ultracentrifugación de sobrenadantes de cultivo o células rotas, los virus pueden concentrarse y quedar disponibles para futuras investigaciones. Esto incluye, entre otras cosas, la determinación microscópica electrónica de partículas de virus. Análisis posteriores de proteínas o genomas virales sirven para caracterizar el tipo de virus.